¿Por qué el análisis exploratorio de datos?
Publicado el: 06 de Diciembre del 2020 - Jhonatan Montilla
Cuando comencé a estudiar sobre aprendizaje automático, solía leer blogs y ver videos, en todos estos señalaban que el EDA es uno de los pasos esenciales, esto plantea una pregunta, ¿por qué?. Imagina que planeas comprar algún producto, ¿qué haces primero?. Intenta comprender las características del producto, qué características tiene y qué características se requieren para satisfacer su necesidad con ese producto. De manera similar, el análisis exploratorio de datos es una tarea que realiza un individuo para familiarizarse con los datos.
Definición :
Es un enfoque para el análisis de datos que pospone las suposiciones habituales sobre qué tipo de modelo siguen los datos con el proceso más natural de permitir que los datos en sí mismos revelen su estructura y modelo subyacentes. EDA no es una mera colección de técnicas. EDA es una filosofía sobre cómo analizamos un conjunto de datos, qué buscamos, cómo nos vemos y cómo interpretamos. Es cierto que EDA utiliza en gran medida la colección de técnicas que llamamos "gráficos estadísticos", pero no es gráficos estadísticos.
¿Qué es el análisis de datos exploratorios (EDA)?
El análisis exploratorio de datos (EDA) es un enfoque / filosofía para el análisis de datos que emplea una variedad de técnicas (principalmente gráficas) para que:
- Los datos se pueden usar en algoritmos de aprendizaje automático.
- EDA le ayuda a elegir los algoritmos más adecuados para su conjunto de datos.
- Le permite determinar la característica potencialmente ideal para el algoritmo de aprendizaje automático
- Maximizar el conocimiento de un conjunto de datos
- Descubrir la estructura subyacente
- Extraer variables importantes (características)
- Detectar valores atípicos y anomalías en el conjunto de datos.
- Determine la configuración óptima de los factores.
Análisis de datos exploratorios explicado utilizando un conjunto de datos de muestra:
Realizaremos un ejemplo con un conjunto de datos de supervivencia de Haberman, que está disponible en el repositorio de aprendizaje automático de la UCI, y trataré de obtener la mayor cantidad de información valiosa del conjunto de datos mediante EDA.
Antes de comenzar será necesario descargar el conjunto de datos a través del siguiente enlace al repositorio. Y se deben importar las librerías necesarias.
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
import numpy as np
import warnings
warnings.filterwarnings('ignore')
haberman = pd.read_csv("haberman.csv")
print (haberman.shape)
(306, 4)
- El conjunto de datos contiene 306 puntos de datos
- El conjunto de datos contiene 4 características
print(haberman.columns)
Index(['age', 'year', 'nodes', 'status'], dtype='object')
haberman['status'].value_counts()
1 225 2 81 Name: status, dtype: int64
- El conjunto de datos está desequilibrado
- Hay dos clases
- Clase 1: el paciente sobrevivió
- Clase 2: el paciente no sobrevivió
- La clase 1 tiene 225 puntos de datos y la clase 2 tiene 81 puntos de datos
Análisis univariado: PDF¶
sns.FacetGrid(haberman, hue="status", size=5) \
.map(sns.distplot, "age") \
.add_legend();
plt.ylabel('% of patient ')
plt.title("Histogram of age")
plt.show();
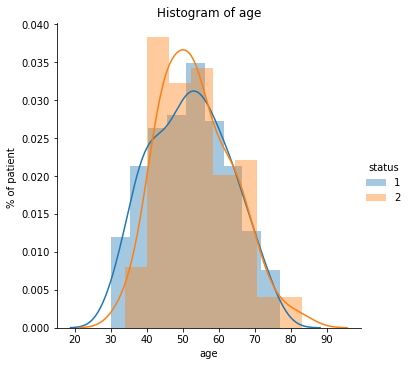
- Según este gráfico, no podemos concluir porque se superponen estrechamente entre sí
sns.FacetGrid(haberman, hue="status", size=5) \
.map(sns.distplot, "year") \
.add_legend();
plt.ylabel('% of patient ')
plt.title("Histogram of year")
plt.show();
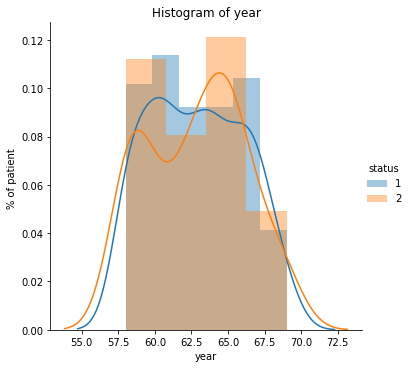
- Según este gráfico, no podemos concluir porque se superponen estrechamente
sns.FacetGrid(haberman, hue="status", size=5) \
.map(sns.distplot, "nodes") \
.add_legend();
plt.ylabel('% of patient ')
plt.title("Histogram of nodes")
plt.show();
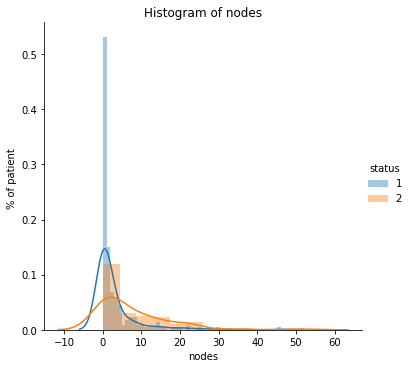
-
Según este gráfico, no podemos completarlo con precisión, pero
-
Nodo 0–4 50 +% de probabilidad de sobrevivir, así como (10–15)% de probabilidad de morir también, no puedo estar seguro
-
Nodo 4-60: la probabilidad de supervivencia es significativamente menor
Análisis Univariante: CDF¶
survived = haberman.loc[haberman["status"] == 1]
not_survived = haberman.loc[haberman["status"] == 2]
label = ["pdf of class 1", "cdf of class 1","pdf of class 2", "cdf of class 2"]
counts, bin_edges = np.histogram(survived["nodes"], bins=10, density = True)
pdf = counts/(sum(counts))
cdf = np.cumsum(pdf)
plt.title("pdf VS cdf for nodes")
plt.xlabel('nodes')
plt.ylabel('% of patient ')
plt.plot(bin_edges[1:], pdf)
plt.plot(bin_edges[1:], cdf)
plt.legend(label)
counts, bin_edges = np.histogram(not_survived["nodes"], bins=10, density = True)
pdf = counts/(sum(counts))
cdf = np.cumsum(pdf)
plt.plot(bin_edges[1:], pdf)
plt.plot(bin_edges[1:], cdf)
plt.legend(label)
plt.show();
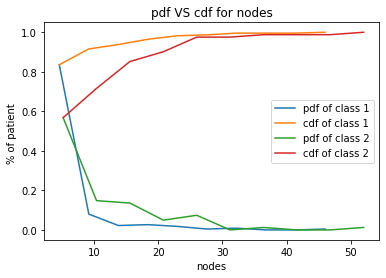
- El paciente que tiene más de 47 nodos no puede sobrevivir
survived = haberman.loc[haberman["status"] == 1]
not_survived = haberman.loc[haberman["status"] == 2]
label = ["pdf of class 1", "cdf of class 1","pdf of class 2", "cdf of class 2"]
counts, bin_edges = np.histogram(survived["year"], bins=10, density = True)
pdf = counts/(sum(counts))
cdf = np.cumsum(pdf)
plt.title("pdf VS cdf for year")
plt.xlabel('year')
plt.ylabel('% of patient ')
plt.plot(bin_edges[1:], pdf)
plt.plot(bin_edges[1:], cdf)
plt.legend(label)
counts, bin_edges = np.histogram(not_survived["year"], bins=10, density = True)
pdf = counts/(sum(counts))
cdf = np.cumsum(pdf)
plt.plot(bin_edges[1:], pdf)
plt.plot(bin_edges[1:], cdf)
plt.legend(label)
plt.show();
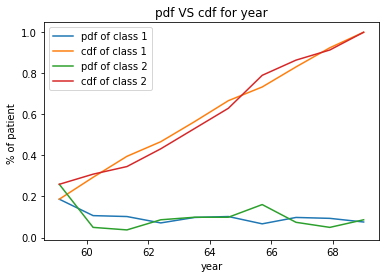
survived = haberman.loc[haberman["status"] == 1]
not_survived = haberman.loc[haberman["status"] == 2]
label = ["pdf of class 1", "cdf of class 1","pdf of class 2", "cdf of class 2"]
counts, bin_edges = np.histogram(survived["age"], bins=10, density = True)
pdf = counts/(sum(counts))
cdf = np.cumsum(pdf)
plt.title("pdf VS cdf for age")
plt.xlabel('age')
plt.ylabel('% of patient ')
plt.plot(bin_edges[1:], pdf)
plt.plot(bin_edges[1:], cdf)
plt.legend(label)
counts, bin_edges = np.histogram(not_survived["age"], bins=10, density = True)
pdf = counts/(sum(counts))
cdf = np.cumsum(pdf)
plt.plot(bin_edges[1:], pdf)
plt.plot(bin_edges[1:], cdf)
plt.legend(label)
plt.show();
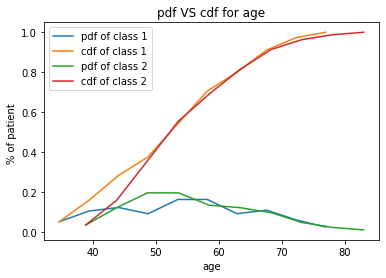
- (16-17)% de los pacientes que tienen menos de 38 años tienen la posibilidad de sobrevivir
Análisis univariante: Diagrama de caja¶
sns.boxplot(x='status',y='nodes', data=haberman)
plt.title("Boxplot of nodes")
plt.show()
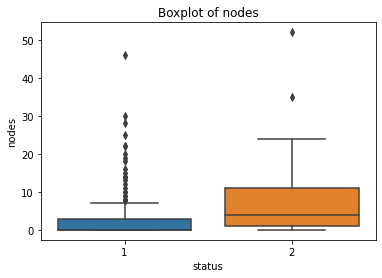
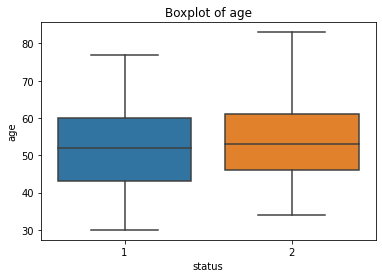
sns.boxplot(x='status',y='age', data=haberman)
plt.title("Boxplot of age")
plt.show()
sns.boxplot(x='status',y='year', data=haberman)
plt.title("Boxplot of year")
plt.show()
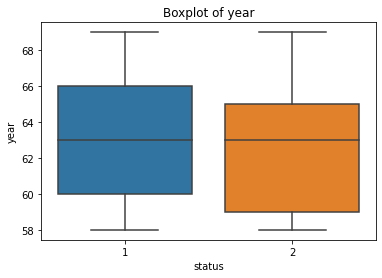
- 50% de probabilidad de sobrevivir si los nodos son menos de tres y
- hay un 15-20% de probabilidad de que no sobrevivan los nodos de identificación son> 3 y <5
- si el nodo tiene> 5 posibilidades de sobrevivir es muy raro
Análisis univariante: gráficos de Voilin¶
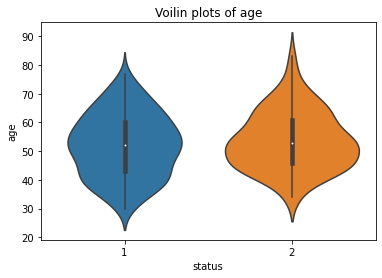
sns.violinplot(x = "status", y = "age", data = haberman)
plt.title("Voilin plots of age")
plt.show()
sns.violinplot(x = "status", y = "nodes", data = haberman)
plt.title("Voilin plots of nodes")
plt.show()
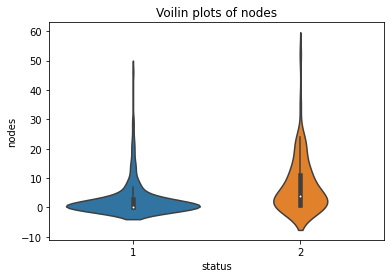
haberman.plot(kind='scatter', x='age', y='year') ;
plt.title("scatter plots of age")
plt.show()
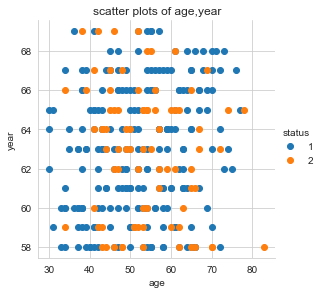
sns.set_style("whitegrid");
sns.FacetGrid(haberman, hue="status", size=4) \
.map(plt.scatter, "age", "year") \
.add_legend();
plt.title("scatter plots of age,year")
plt.show();
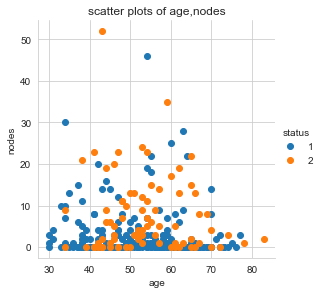
sns.set_style("whitegrid");
sns.FacetGrid(haberman, hue="status", size=4) \
.map(plt.scatter, "age", "nodes") \
.add_legend();
plt.title("scatter plots of age,nodes")
plt.show();
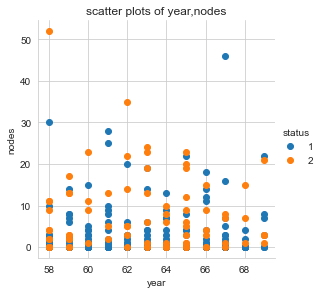
sns.set_style("whitegrid");
sns.FacetGrid(haberman, hue="status", size=4) \
.map(plt.scatter, "year", "nodes") \
.add_legend();
plt.title("scatter plots of year,nodes")
plt.show();
plt.close();
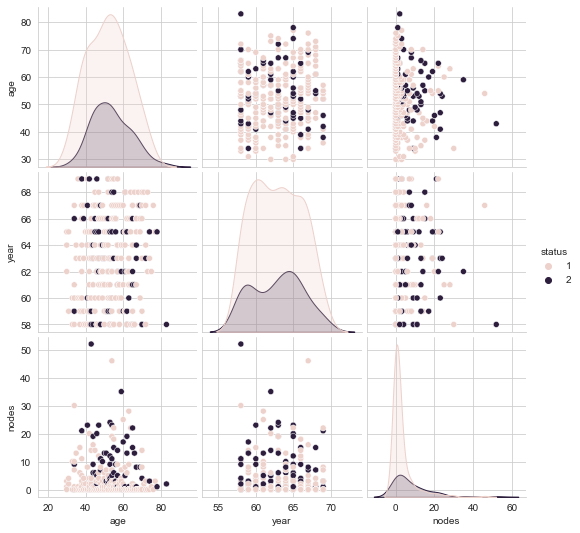
sns.set_style("whitegrid");
sns.pairplot(haberman, hue="status", vars=['age', 'year','nodes']);
plt.show()